TP19 : Le contrôle de l’inflammation : Les médicaments anti-inflammatoires
La réaction inflammatoire fait intervenir des leucocytes qui jouent un rôle précis dont celui de produire des médiateurs de l’inflammation (cytokines, histamine et prostaglandine). Ces molécules induisent des effets différents et nécessaires au bon déroulement de la réaction inflammatoire.
Les symptômes stéréotypés de la réaction inflammatoire (rougeur, gonflement, chaleur et douleur) correspondent aux résultats de l’action de ces différents médiateurs de l’inflammation.
Les médicaments de la classe des anti-inflammatoires (comme leur nom l’indique) permettent de réduire les symptômes de l’inflammation en particulier le gonflement pouvant provoquer des gênes mais surtout la douleur.
Problème : Comment agissent ces médicaments? A quel niveau agissent-ils pour réduire les symptômes de l’inflammation?
[important]
Consigne :
A partir de l’analyse des documents et de l’exploitation du logiciel Rastop, expliquer comment l’aspirine ou l’ibuprofène permettent de réduire les symptômes de l’inflammation.
La réponse complète doit montrer :
- A quel niveau de la réaction inflammatoire agit le médicament.
- Comment agit le médicament au niveau moléculaire (des captures d’images issues du logiciel Rastop doivent l’illustrer).
- Donner les effets indésirables éventuels de ces médicaments.
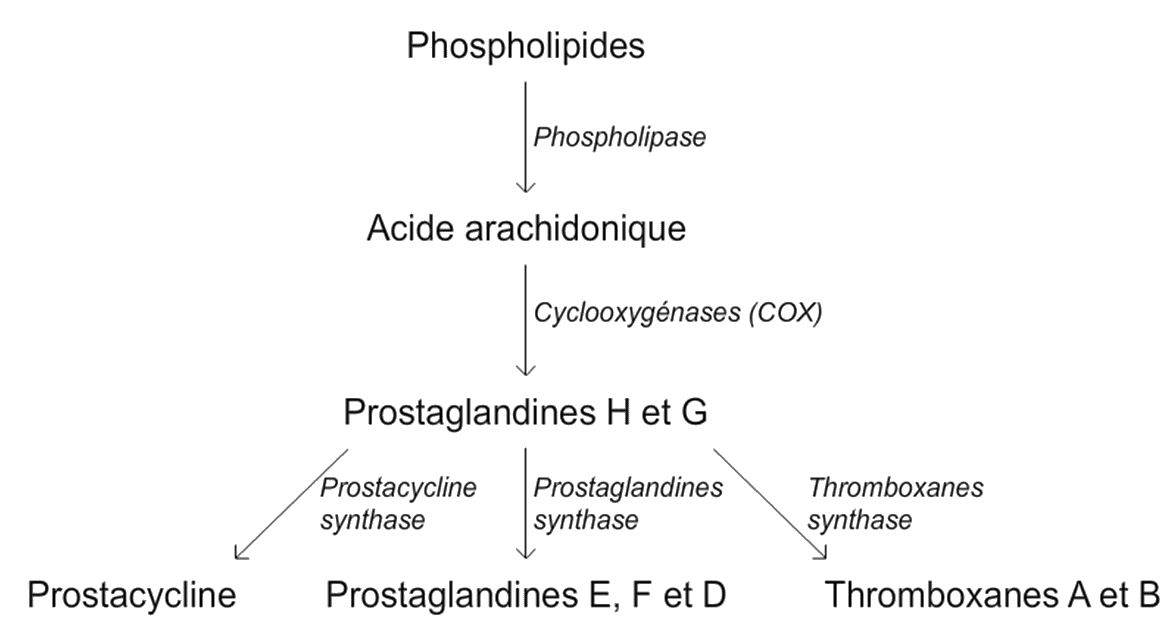
Document 1 : La chaîne de biosynthèse des prostaglandines
Parmi les molécules synthétisées lors de la réaction inflammatoire aiguë, certaines prostaglandines provoquent une vasodilatation et une augmentation de la perméabilité vasculaire, et contribuent ainsi à l’apparition des symptômes inflammatoires. Les étapes de la synthèse des prostaglandines à partir de molécules de la membrane d’une cellule sécrétrice sont représentées sur le document ci-contre.
NB : Chaque transformation chimique ne peut se produire spontanément : chacune dépend l’activité d’une enzyme spécifique.
Document 2 : Les conditions de synthèse de la cyclo-oxygénase (COX) dans les monocytes ou granulocytes
Des chercheurs travaillant sur la réaction inflammatoire se sont intéressés à l’enzyme cyclo-oxygénase (= COX).
On fait incuber un nombre défini de monocytes et de granulocytes en présence d’une concentration de 10 µg/mL de LPS (molécule de la paroi de nombreuses bactéries) pendant différents temps : 0, 1, 2,5 et 4,5 heures. On traite ensuite la culture de manière à récupérer le cytoplasme des cellules, et on effectue une électrophorèse destinée à séparer les molécules de COX des autres protéines cytoplasmiques. La coloration des protéines COX donne les résultats suivants :
| Temps en heures | ||||
|
0 |
1 |
2,5 |
4,5 |
|
|
COX isolée : sa quantité est proportionnelle à la dimension et à l’intensité des taches colorées |
||||
Séparation des enzymes COX du cytoplasme de granulocytes et de monocytes après action du LPS à 10 µg/ml pendant différentes durées – D’après http://www.futura-sciences.com
Document 3 : Mode d’action moléculaire des enzymes de l’ibuprofène et de l’aspirine
 3a. Généralités sur le mode d’action moléculaire des enzymes
3a. Généralités sur le mode d’action moléculaire des enzymes
- Les enzymes sont des protéines constituées de centaines d’acides aminés.
- Pour agir, l’enzyme doit rentrer en contact avec la molécule de substrat qui lui est spécifique pour former un complexe enzyme-substrat. Cette liaison avec la molécule de substrat est suivie de la libération des produits de la réaction. Ce contact s’établit au niveau d’une zone particulière de l’enzyme, zone en creux et complémentaire de forme d’une partie de la molécule de substrat que l’on nomme le site actif. Le site actif est constitué de quelques acides aminés qui assurent une liaison temporaire avec le substrat spécifique ce qui permet le déroulement de la réaction.
3b : Données générales sur le fonctionnement des cyclo-oxygénases
La cyclo-oxygénase (COX) est une enzyme qui permet la formation de prostaglandines à partir de l’acide arachidonique.
Normalement, l’acide arachidonique, libéré des membranes, s’engage dans le site actif de la cyclo-oxygénase par un canal (une cavité dans la molécule) et établit une liaison avec un de ses acides aminés, la Tyrosine en position 385. La transformation de l’acide arachidonique en prostaglandine commence alors. Le canal est délimité par les acides aminés Tyrosine en position 355 et arginine en position 120.
3b. Visualisation du complexe COX-acide arachidonique avec le logiciel RASTOP
Charger le complexe Enzyme-substrat « cox_acide_arachidonique.pdb » (la COX est nommée chaine A ; l’acide arachidonique est appelé ACD 700 ) et utiliser la fiche technique du logiciel pour mettre en évidence judicieusement certaines parties des molécules affichées.
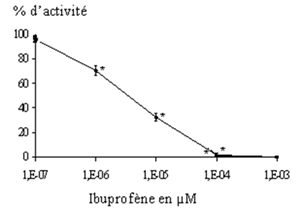 Document 4 : Influence de l’ibuprofène et de l’aspirine sur l’activité de la COX
Document 4 : Influence de l’ibuprofène et de l’aspirine sur l’activité de la COX
4a : Données expérimentales
On mesure de l’activité de l’enzyme COX en présence de concentrations croissantes d’ibuprofène (10-7 à 10-3 µM).
En absence d’ibuprofène, on mesure l’activité d’une quantité « Q » de COX et on lui attribue la valeur 100% d’activité. On ajoute ensuite à la même quantité « Q » de COX une quantité « q » d’ibuprofène, puis on mesure l’activité de l’enzyme (c’est à dire la synthèse de prostaglandine à partir de l’acide arachidonique).
(http://www.futura-sciences.com/)
4b : Données fournies par les études de biologie moléculaire
Les AINS (Anti-inflammatoires non stéroïdiens) peuvent s’engager dans ce canal et établir une transformation de la tyrosine 385. Dans ce cas, l’acide arachidonique ne peut plus s’y loger et par conséquent, la synthèse des prostaglandines est inhibée.
Des études de biologie moléculaire ont montré que la molécule d’ibuprofène et l’aspirine se fixent sur les acides aminés 120 (Arginine) de la COX et de ce fait interagit avec les acides aminés 385 (tyrosine) et 530 (sérine) de l’enzyme .
4b : Visualisation des complexes COX-AINS avec le logiciel RASTOP
Charger le complexe Enzyme-substrat «cox_ac_arachidonique.pdb» et le complexe Enzyme-AINS « cox_aspirine.pdb» ou « cox_ibuprophene.pdb » et utiliser la fiche technique du logiciel pour mettre en évidence judicieusement certaines parties des molécules affichées.